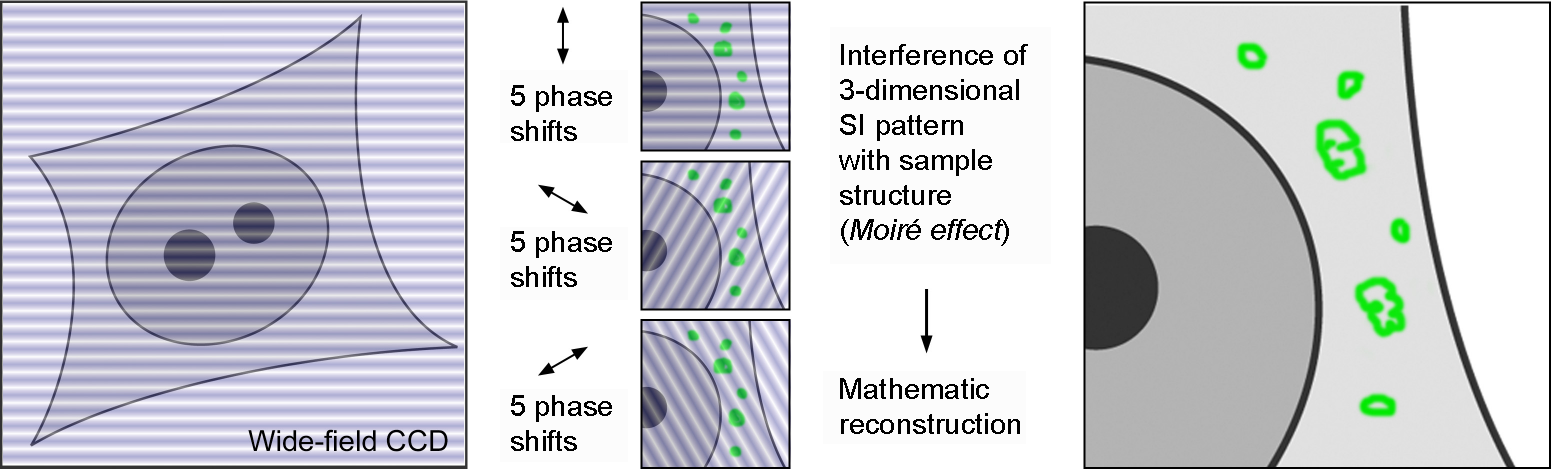
I microscopi a luce strutturata sfruttano le frange di Fourier, ottenute proiettando sul campione una griglia di linee opache/trasparenti, per ricostruire tramite vari algoritmi un'immagine simil-confocale, ma con una risoluzione nella dimensione Z molto superiore. In poche parole, il risultato sono immagini prive dei contributi delle parti fuori fuoco del campione. A causa degli algoritmi usati, che si basano su operazioni sui pixel in scala di grigio, l'immagine finale non può che essere in bianco e nero e solo successivamente alla ricostruzione sarà possibile ricolorarla, perdendo tuttavia una buona parte del range dinamico (aka: immagine solitamente più scura del reale).

Il sistema può essere utilizzato sia in fluorescenza, che in riflessa.
La luce utilizzata DEVE essere incoerente (non polarizzata). Quindi si possono usare tranquillamente sia le vecchie alogene che i nuovi led, magari a banda singola (fluorescenza). Un sistema laser è utilizzabile, ma la luce dovrà essere depolarizzata. In questo caso è addirittura possibile ottenere le linee della griglia facendo interferire le lunghezze d'onda di due laser.
Il sistema che attualmente sembra dare i migliori risultati è però l'utilizzo di un SLM (spatial light modulator, o DMD), ovvero un sistema di microspecchi, usato spesso nei proiettori moderni, controllati dal software che proiettano sul campione solo la luce riflessa dagli specchi in una certa posizione.

Il mio sistema si basa su un reticolo di Ronchi da 600 linee/pollice che fornisce il pattern della griglia, sul modello dello Zeiss Apotome, il sistema a luce strutturata Zeiss che tuttavia non è in grado di ruotare la griglia ad angoli diversi. La griglia dell'Apotome è mossa da un motore piezo e le immagini (3 o 5), vengono registrate da una videocamera ad altissima sensibilità che permette al programma di fornire un'immagine in tempo reale delle sezioni, sempre in bianco e nero. In poche parole, il sistema Zeiss ha tempi di acquisizione molto bassi grazie allo spostamento piezo, ad una fotocamera molto performante e al ridotto numero di foto da ricostruire per ogni sezione (3 o 5) contro i 15 frame del mio sistema (5 frame per 3 angoli).
Ok, il mio sistema è lento ad acquisire (circa 15-20 secondi a sezione), ma alla fine anche l'Apotome non potrebbe essere utilizzato su campioni vivi. I sistemi veloci devono essere basati su un SLM o interferenza di raggi laser.... ma diciamo che nel mio piccolo posso accontentarmi!
Le 15 immagini con la griglia sovraimposta vengono "stackate" con ImageJ in un unico file tiff multi-immagine e processate con un programma open-source, il SIMtoolbox, che offre molti algoritmi di ricostruzione e opzioni per adattarlo al proprio sistema.
Era da quasi un anno che aspettavo che il fresatore mi preparasse i pezzi necessari, che sono stati progettati in CAD e realizzati in alluminio e ottone:
Il primo prototipo è stato realizzato con i tristi resti di un micromanipolatore, un micrometro per la traslazione del reticolo, e qualche pezzetto di alluminio. Le prove fatte con questo rudimentale accrocchio hanno dato dei risultati molto interessanti, il che ha fatto partire il progetto definitivo. Ecco una delle primissime immagini di alghe filamentose prima (con griglia sovraimposta) e dopo la ricostruzione, con soli 3 frame coinvolti:



Ora una sezione di fusto di muschio, ricostruita da 15 immagini, sempre con l'accrocchio sperimentale iniziale, movimenti sprecisi causano difetti nella ricostruzione, ma per rendere l'idea va bene:
immagini in fluorescenza UV


Come fonte UV sto usando il più potente led UV in circolazione, della Nichia, piuttosto raro e costoso, il NVSU333A. Lo uso a 1/10 della sua potenza e già così il campione ne risente velocemente....
La fotocamera è una Basler Scout scA1400-17gm con trigger esterno e abbastanza sensibile (efficienza quantica 58%)... la velocità di acquisizione è in grandissima parte legata al tempo di esposizione.
I vari pezzi, finalmente assemblati pochi giorni fa:

Un servo ruota la griglia e uno stepper da 200 step/rev collegato ad un micrometro (sempre del vecchio micromanipolatore) da 200 divisioni da 1 micron per giro ne permette la traslazione con precisione micrometrica. Quindi posso regolare con precisione la traslazione della griglia, uno step = micron.
Il sistema di controllo di tutto il microscopio si basa su due Arduino, due driver per gli stepper del tavolo motorizzato, uno shield per gli stepper della messa a fuoco (ridotto 5.18:1), per il motore che muove la griglia e per il servo. La fotocamera, i motori, la reflex Canon e il monitor "di servizio" sopra la testata sono alimentati da un alimentatore stabilizzato da 10A 12V e alcuni step-down.
I programmi del SIM e per gli stack sono pilotati tramite touch screen. Tavolo e messa a fuoco sono comandati da joystick e pulsanti sul controller esterno.

Ecco un primo video del funzionamento. Tramite il touch screen seleziono la sezione singola, servo e motore muovono il reticolo focalizzato sul piano di fuoco posteriore dell'obiettivo e la fotocamera registra i frame, sempre controllata da Arduino e alla fine mi trovo con le 15 immagini da ricostruire in una cartella:
[youtubenew]ViBGOUhJGXg[/youtubenew]
Il programma prevede l'acquisizione di più sezioni verticali per una ricostruzione tridimensionale dei soggetti. Per ora sto testando solo le sezioni singole.
Prossimamento posterò gli sviluppi e le prime immagini ricostruite. Al momento devo ancora trovare le distanze giuste di traslazione della griglia.
 Iscrivetevi al canale YouTube di Binomania!
Iscrivetevi al canale YouTube di Binomania!

